Planejamento de análogos do GC376
- InfoVid-19 Winx

- 22 de jun. de 2021
- 8 min de leitura
ATENÇÃO: o conteúdo desse post está intimamente relacionado com o post anterior. Recomendamos que seja feita sua leitura para melhor compreensão dos termos e discussões. Obrigada!
No post anterior, nós conhecemos o inibidor GC376, um possível candidato a fármaco contra a COVID-19. Falamos sobre suas principais características farmacocinéticas e sobre suas propriedades físico-químicas e farmacodinâmicas. Descobrimos que o GC376 é um ótimo inibidor, mas será que conseguimos melhorá-lo? Pois bem, foi o que tentamos fazer: desenvolver análogos do GC376 para aumentar seu potencial inibitório e outras propriedades.
O planejamento de análogos é essencial para que compostos com potencial atividade biológica possam ser desenvolvidos com mais eficiência e seletividade e menos toxicidade e efeitos colaterais. (AVER, et al., 2015). E esse processo de planejamento acontece da seguinte maneira:
O planejamento e desenvolvimento de novos fármacos é realizado por uma área da Química Farmacêutica Medicinal. Segundo a União Internacional de Química Pura e Aplicada (IUPAC) envolve áreas inter e multidisciplinares do conhecimento, tais como a Química, Ciências Médicas, Ciências Farmacêuticas e Ciências Biológicas e uma das técnicas mais utilizadas para esse processo é a química computacional (PINZI; RASTELLI, 2019). Uma técnica computacional bastante utilizada é o docking molecular, que utilizamos no post anterior para observar como o ligante GC376 se encaixa no seu alvo molecular. Esse e outros métodos de avaliação de desempenho e métodos baseados na similaridade dos ligantes tornaram possíveis a realização de estratégias de modificação molecular com objetivo de otimizar características farmacodinâmicas e/ou farmacocinéticas, para que possíveis análogos candidatos possam ser identificados. O sucesso no planejamento está relacionado com o entendimento do alvo molecular, pois a partir dele podemos testar possíveis ligantes e selecionar características estruturais mais adequadas (SANTOS, 2016).
Desse modo, nessa etapa foram propostos 3 análogos, que denominamos de InfoVid 1, 2 e 3, baseados na estrutura do inibidor GC376 e no que sabemos sobre sua interação com o alvo molecular, visando aprimorar suas propriedades farmacocinéticas e/ou farmacodinâmica.
ONDE ALTERAR?
O principal objetivo da formação de análogos é “melhorar” o desempenho do ligante por meio de mudanças em sua estrutura, as quais interferem diretamente em suas características físico-químicas. Essas características podem ser: flexibilidade, polaridade e estabilidade. O conhecimento dessas características é essencial para a formulação de um fármaco eficaz no tratamento.
Flexibilidade: Capacidade da molécula se "dobrar" e mudar seu formato e quanto menor, melhor, já que quanto mais fixa a sua posição, melhor o encaixe (PEREIRA, 2007).
Polaridade: Carga elétrica da molécula, interfere diretamente a relação do fármaco com as barreiras do organismo e com suas ligações e quando menor, melhor, pois nossas barreiras (membranas) são, em sua grande maioria, lipoproteicas (PEREIRA, 2007).
Estabilidade: A capacidade do fármaco de se manter em seu formato original e não perder suas características e quanto maior essa capacidade, melhor! Mais estabilidade = melhor encaixe (PEREIRA, 2007).
E ao escolher qual propriedade melhorar, devemos conhecer mais da estrutura do inibidor e a sua complexação com a Mpro, e assim, saber quais serão os locais adequados para esta mudança. Para localizarmos os pontos específicos, sem prejudicar a ligação com o receptor ou sua estrutura de encaixe, utilizamos o gráfico Ligplot (figura 1), obtido na plataforma PDBSum.

Figura 1: Gráfico Ligplot do inibidor GC376 complexado com o bolsão catalítico da Mpro. As linhas pontilhadas em verde demonstram ligações de hidrogênio feitas entre os aminoácidos da Mpro e os átomos do ligante, enquanto os "sóis" vermelhos representam aminoácidos próximos que interagem apenas de forma hidrofóbica com o ligante. A linha contínua roxa entre o C28 do ligante e o átomo de enxofre da Cys 145 representa uma ligação covalente. Fonte: PDBsum (ID 7D1M)
As ligações e interações que podemos ver na figura 1 acima, não podem ser modificadas, então o desafio é encontrar pontos estratégicos para realizar alterações que não irão comprometer o que o inibidor já tem de bom. Se houver uma mudança no encaixe, a interação pode se tornar mais instável ou até mesmo ser impedida, comprometendo a afinidade. Pensando nisso, encontramos alguns pontos no inibidor GC376 que poderíamos alterar: 2 de seus oxigênios e dois carbonos que não realizavam nenhuma interação (figura 2).
Agora podemos propor os análogos mais adequados e prever suas propriedades, com o intuito de verificar se houve melhora nas mesmas.
AS ALTERAÇÕES
As alterações realizadas são as seguintes:

Figura 2: locais de modificação de cada análogo. Fonte: Próprio autor.
INFOVID 1: Adição de um grupo hidroxila no anel aromático do inibidor GC37
Para desenvolvermos o InfoVid 1 primeiro procuramos um aminoácido próximo ao inibidor que não estivesse realizando interação com ele. Encontramos uma prolina (168) e averiguamos que o nitrogênio dela está a 4.31 Å de distância do carbono 2 do ligante (figura 3) ou seja, há espaço e proximidade o suficiente para uma ligação de hidrogênio. Pensando nisso, adicionamos uma hidroxila (OH) a esse carbono, permitindo que mais uma ligação fosse feita entre o inibidor e o sítio catalítico, permitindo com que tivesse mais um ponto de contato com a protease viral.

Figura 3: Distância entre o carbono 2 do inibidor GC376 e o nitrogênio da Pro168. Fonte: ViewerLite.
INFOVID 2: Troca de oxigênios por carbonos;
Para o segundo e terceiro análogos a intenção era de melhorar suas características farmacocinéticas, então utilizamos o gráfico ligplot e alteramos algumas características: no segundo análogo trocamos os oxigênios que não estavam fazendo ligações (8 e 18) por carbonos para diminuir a polaridade da molécula. Só temos um problema: aumentar o número de carbonos aumenta a flexibilidade, então também adicionamos uma ligação dupla entre um dos novos carbonos (substituinte do Oxigênio 8) e o carbono 7 que já estava lá.
INFOVID 3: Homologação ou adição à cadeia carbônica;
Com o terceiro análogo a intenção foi a mesma do anterior: aumentar a lipofilicidade. Dessa vez a ideia foi de aumentar a cadeia carbônica por meio da edição de grupos CH2. O local escolhido foi entre o carbono 13 e o carbono 14 (figura 1). Sempre importante lembrar que esse "braço" da molécula não realizava ligações com o sítio ativo e por isso as alterações não causaram mudança na ligação inibidor-protease. Por meio da pesquisa no banco de dados SwissADME, conseguimos comparar a diferença na lipofilicidade entre diferentes moléculas e calculamos 3 valores para a quantidade de grupos CH2 que adicionaríamos, sendo esse valor o n (n=1, n=2, n=3), e averiguamos qual seria o valor mínimo para se obter o máximo valor de logP. Concluímos que número mínimo seria de 2, o que representa o acréscimo de (CH2)2 ou C2H4 na cadeia carbônica deste análogo.
METODOLOGIA
Utilizamos o programa Marvin 21.8 para analisar os estados de protonação (carga dos átomos), tautomerismo (equilíbrio) e estereoquímica (formato) dos análogos propostos. Em seguida, realizamos uma otimização na estrutura do ligante por meio da busca conformacional, onde ajustamos o campo de força (MMFF94) no programa Avogadro, para posteriormente realizar os cálculos de docking molecular e por fim, avaliar as propriedades ADME-Tox.
Abaixo vamos apresentar seus estados protonados, seu tautomerismo, estereoquímica e também apresentar e discutir as suas propriedades ADMETox, drug-likeness e as interações proteína-ligante em comparação com o inibidor original.
INFOVID 1

Figura 4: Local de alteração do InfoVid 1. Fonte: Próprio autor.
Esse análogo apresentou apenas uma forma protonada principal (98,09%) e também apenas um tautômero principal (98,09%). Nas figuras abaixo podemos ver o análogo (figura 4), sua principal forma protonada e seu principal tautômero (figura 5).
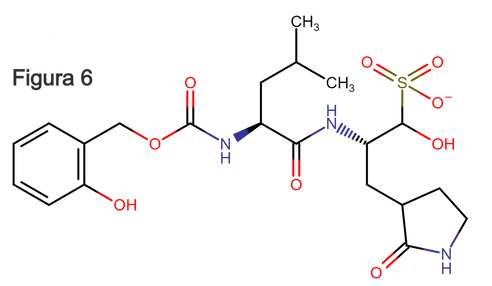
Figura 5: InfoVid 1. A adição do grupo OH no anel aromático do inibidor permite uma ligação de hidrogênio com a prolina. Figura 6: Principal forma protonada (98,09%) e também principal tautômero (98,09%) do InfoVid 1 em pH biológico (pH 7.4). Fonte: Marvin
Graças à adição, houve também algumas alterações nas propriedades cinéticas e dinâmicas do fármaco (tabela 1):

Tabela 1: características farmacocinéticas e dinâmicas do InfoVid 1. Fonte: SwissADME; ADMETlab.
O análogo apresenta menor lipofilicidade (-2,781), um fator negativo, o que pode acarretar na diminução de sua absorção, mas outras características tiveram diminuição no valor predito e representam melhoras no candidato a fármaco: ligação com proteínas plasmáticas (o que aumenta a sua disponibilidade na corrente sanguínea), capacidade de bloquear a hERG, sensibilização da pele e hepatotoxicidade. Por outro lado 2 fatores negativos aumentaram: a mutagenicidade (capacidade de causar mutações) e a LD50 (dose necessária para matar 50% da população em teste), e enquanto o aumento da última é considerável (70,8 mg/kg) e animador, a primeira pode causar preocupação, apesar de um aumento de apenas 0,006. A meia vida está mais curta, com um valor de 47 minutos, ou seja, seu tempo de ação está reduzido. Quanto a capacidade de inibir ou servir de substrato para enzimas da CYP, não houveram mudanças.
A fármaco-similaridade desse análogo foi:
Lipinski- 3 violações: MW>500, NorO>10, NHorOH>5
Ghose- 1 violações: MW>480
Veber- 2 violações: Rotors>10, TPSA>140
Egan- 1 violações: TPSA>131.6
Muegge- 2 violações: TPSA>150, H-don>5
Não apresentando nenhuma fármaco-similaridade.
INFOVID 2

Figura 7: Local de alteração do InfoVid 2. Fonte: Próprio autor.
Nas figuras abaixo podemos ver o análogo InfoVid 2 (figura 8), sua principal forma protonada (figura 9), seu principal tautômero (figura 10).
Figura 8: infovid 2. A substituição dos oxigênios 8 e 18 por carbonos e a formação de uma ligação dupla entre o novo carbono e o carbono 7 deixam a molécula menos polar e menos flexível. Figura 9: Principal forma protonada (94,81%) do análogo 2 em pH biológico (pH 7.4). Figura 10: Principal tautômero com 95% de distribuição. Fonte: Marvin
As suas propriedades ficaram as seguintes (tabela 2):

Tabela 2: propriedades do InfoVid 2. Fonte: SwissADME; ADMETlab
O InfoVid 2 apresentou um leve aumento da sua lipofilicidade e sua absorção gastrointestinal (G.I) mesmo aumentando consideravelmente, ainda é baixa. O análogo também se liga mais facilmente às proteínas plasmáticas, baixa probabilidade de bloquear a hERG, moderada capacidade de sensibilização da pele, alta hepatotoxicidade, meia vida de 1 hora e 25 minutos e uma LD50 duas vezes maior que o GC376 original. Sobre seu metabolismo, o análogo 2 demonstrou capacidade de ser substrato da CYP3A4.
A fármaco-similaridade desse infovid foi:
Lipinski- 0 violações
Ghose- 2 violações: MW>480, MR>130
Veber- 2 violações: Rotors>10, TPSA>140
Egan- 1 violação: TPSA>131.6
Muegge- 1 violação: TPSA>150
Apresentando farmaco-similaridade segundo os critérios de Lipinski com 0 violações.
INFOVID 3

Figura 11: Local de alteração do InfoVid 3. Fonte: Próprio autor.
Por meio da pesquisa no banco de dados SwissADME, conseguimos comparar a diferença na lipofilicidade entre diferentes moléculas e calculamos 3 valores para a quantidade de grupos CH2 que adicionaríamos, sendo esse valor o n (n=1, n=2, n=3), e averiguamos qual seria o valor mínimo para se obter o máximo valor de logP. Concluímos que número mínimo seria de 2, o que representa o acréscimo de (CH2)2 ou C2H4 na cadeia carbônica deste análogo. Nas figuras abaixo podemos ver o análogo (figura 13), sua principal forma protonada e seu principal tautômero (figura 14).
Figura 12: InfoVid 3. A adição de grupos CH2 aumentam a cadeia carbônica aumentando a lipossolubilidade da molécula. Figura 13: Principal forma protonada e também principal tautômero (100%) do InfoVid 3 em pH biológico (pH 7.4). Fonte: Marvin
As propriedades desse análogo (tabela 3):

Tabela 3: propriedades do InfoVid 3. Fonte: SwissADME; ADMETlab
O Log P do InfoVid 3 é mais que o dobro do GC376 original, apresentando um resultado positivo na nossa tentativa de aumentar a lipofilicidade. Esse análogo também possui maior capacidade tóxica com um aumento na hepatotoxicidade, mutagenicidade, capacidade de bloquear a hERG e capacidade de sensibilizar a pele. O InfoVid 3 apresentou maior capacidade de ligar-se às proteínas plasmáticas, meia vida de aproximadamente 1 hora e baixa absorção G.I. Sobre o seu metabolismo, a molécula é uma possível inibidora da CYP3A4 e um possível substrato da CYP2C9.
A fármaco-similaridade desse análogo foi:
Lipinski- 2 violações: MW>500, NorO>10
Ghose- 2 violações MW>480, MR>130
Veber- 2 violações: Rotors>10, TPSA>140
Egan- 1 violação: TPSA>131.6
Muegge- 2 violações: TPSA>150, Rotors>15
Não apresentando nenhuma fármaco-similaridade.
REFERÊNCIAS
AVER, G. M., Kreutz, O. C., & Suyenaga, E. S. (2015). MÉTODOS DE OBTENÇÃO DE FÁRMACOS SOB A ÓPTICA DA QUÍMICA MEDICINAL. Revista Conhecimento Online, 2, 63–73.
CERA, Tatiane Peixoto et al. Planejamento de Fármacos. Artigo, Revista Científica UNILAGO, USP, São Paulo. Disponível em: http://www.unilago.edu.br/revista/edicaoanterior/Sumario/2013/downloads/2013/PLANEJAMENTO%20DE%20F%C3%81RMACOS.pdf Acesso em 25 de maio de 2021.
GUENGERICH, Peter F. CYTOCHROME P-450 3A4: Regulation and Role in Drug Metabolism, apr.1999. Disponível em: https://doi.org/10.1146/annurev.pharmtox.39.1.1 Acesso em 27 de maio de 2021.
PEREIRA, D. G. Importância do metabolismo no planejamento de fármacos. Química Nova, v. 30, n. 1, p. 171–177, fev. 2007.
PINZI, Luca; RASTELLI, Giulio. Molecular Docking: Shifting Paradigms in Drug Discovery, set. 2019. Disponível em: https://doi.org/10.3390/ijms20184331 Acesso em 25 de maio de 2021.
REGRA dos Cinco e suas extensões para análise de drug-likeness. Gravação de Química Farmacêutica & Medicinal. Youtube: [s. n.], 2020. Disponível em: https://www.youtube.com/watch?v=5xAQuUUkr9c&t=314s. Acesso em: 8 jun. 2021.
SANTOS, L. H. S. dos. Planejamento racional de fármacos aplicado à busca e otimização de inibidores do HIV-1 e Doença de Chagas. 2016. 159f. Tese (Doutorado em Biologia Computacional e Sistemas) - Fundação Oswaldo Cruz, Instituto Oswaldo Cruz, Rio de janeiro, RJ, 2016


















Comentários